References
[ 1 ]
Martin L, Hutchens M, Hawkins C, Radnov A. How much do clinical trials cost? Nat Rev Drug Discov 2017;16(6):381‒2.
link1
[ 2 ]
Moore TJ, Zhang H, Anderson G, Alexander GC. Estimated costs of pivotal trials for novel therapeutic agents approved by the US Food and Drug Administration, 2015‒2016. JAMA Intern Med 2018;178(11):1451‒7.
link1
[ 3 ]
Paul SM, Mytelka DS, Dunwiddie CT, Persinger CC, Munos BH, Lindborg SR, et al. How to improve R&D productivity: the pharmaceutical industry’s grand challenge. Nat Rev Drug Discov 2010;9(3):203‒14.
link1
[ 4 ]
Hopkins AL. Network pharmacology: the next paradigm in drug discovery. Nat Chem Biol 2008;4(11):682‒90.
link1
[ 5 ]
Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet 2009;10(1):57‒63.
link1
[ 6 ]
Giacomotto J, Ségalat L. High-throughput screening and small animal models, where are we? Br J Pharmacol 2010;160(2):204‒16.
link1
[ 7 ]
Mayr LM, Bojanic D. Novel trends in high-throughput screening. Curr Opin Pharmacol 2009;9(5):580‒8.
link1
[ 8 ]
Shoichet BK. Virtual screening of chemical libraries. Nature 2004;432(7019):862‒5.
link1
[ 9 ]
Kitchen DB, Decornez H, Furr JR, Bajorath J. Docking and scoring in virtual screening for drug discovery: methods and applications. Nat Rev Drug Discov 2004;3(11):935‒49.
link1
[10]
LeCun Y, Bengio Y, Hinton G. Deep learning. Nature 2015;521(7553):436‒44.
link1
[11]
Farabet C, Couprie C, Najman L, LeCun Y. Learning hierarchical features for scene labeling. IEEE Trans Pattern Anal Mach Intell 2013;35(8):1915‒29.
link1
[12]
Dahl GE, Yu D, Deng L, Acero A. Context-dependent pre-trained deep neural networks for large-vocabulary speech recognition. IEEE Trans Audio Speech 2012;20(1):30‒42.
link1
[13]
Ding J, Sharon N, Bar-Joseph Z. Temporal modelling using single-cell transcriptomics. Nat Rev Genet 2022;23(6):355‒68.
link1
[14]
Liu S, Trapnell C. Single-cell transcriptome sequencing: recent advances and remaining challenges. F1000 Res 2016;5(1):182.
link1
[15]
Luecken MD, Theis FJ. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol 2019;15(6):e8746.
link1
[16]
Aebersold R, Mann M. Mass spectrometry-based proteomics. Nature 2003;422(6928):198‒207.
link1
[17]
Kim S, Chen J, Cheng T, Gindulyte A, He J, He S, et al. PubChem in 2021: new data content and improved web interfaces. Nucleic Acids Res 2021;49(D1): D1388‒95.
link1
[18]
Bateman A, Martin MJ, Orchard S, Magrane M, Agivetova R, Ahmad S, et al.; Consortium UniProt. UniProt: the universal protein knowledgebase in 2021. Nucleic Acids Res 2021;49(D1):D480‒9.
[19]
Manzoni C, Kia DA, Vandrovcova J, Hardy J, Wood NW, Lewis PA, et al. Genome, transcriptome and proteome: the rise of omics data and their integration in biomedical sciences. Brief Bioinform 2018;19(2):286‒302.
link1
[20]
Shi Y, Prieto PL, Zepel T, Grunert S, Hein JE. Automated experimentation powers data science in chemistry. Acc Chem Res 2021;54(3):546‒55.
link1
[21]
Nam AS, Chaligne R, Landau DA. Integrating genetic and non-genetic determinants of cancer evolution by single-cell multi-omics. Nat Rev Genet 2021;22(1):3‒18.
link1
[22]
Waring MJ, Arrowsmith J, Leach AR, Leeson PD, Mandrell S, Owen RM, et al. An analysis of the attrition of drug candidates from four major pharmaceutical companies. Nat Rev Drug Discov 2015;14(7):475‒86.
link1
[23]
Tunyasuvunakool K, Adler J, Wu Z, Green T, Zielinski M, Žídek A, et al. Highly accurate protein structure prediction for the human proteome. Nature 2021;596(7873):590‒6.
link1
[24]
Ying C, Cai T, Luo S, Zheng S, Ke G, He D, et al. Do transformers really perform badly for graph representation? Adv Neural Inf Process Syst 2021;34(1):28877‒88.
[25]
Seyed Tabib NS, Madgwick M, Sudhakar P, Verstockt B, Korcsmaros T, Vermeire S. Big data in IBD: big progress for clinical practice. Gut 2020;69(8):1520‒32.
link1
[26]
Granda JM, Donina L, Dragone V, Long DL, Cronin L. Controlling an organic synthesis robot with machine learning to search for new reactivity. Nature 2018;559(7714):377‒81. Corrected in: Nature 2019;570:E67‒9.
link1
[27]
Zhong F, Xing J, Li X, Liu X, Fu Z, Xiong Z, et al. Artificial intelligence in drug design. Sci China Life Sci 2018;61(10):1191‒204.
link1
[28]
Zhavoronkov A, Ivanenkov YA, Aliper A, Veselov MS, Aladinskiy VA, Aladinskaya AV, et al. Deep learning enables rapid identification of potent DDR1 kinase inhibitors. Nat Biotechnol 2019;37(9):1038‒40.
link1
[29]
Winter R, Montanari F, Noé F, Clevert DA. Learning continuous and datadriven molecular descriptors by translating equivalent chemical representations. Chem Sci 2019;10(6):1692‒701.
link1
[30]
Zheng S, Rao J, Song Y, Zhang J, Xiao X, Fang EF, et al. PharmKG: a dedicated knowledge graph benchmark for bomedical data mining. Brief Bioinform 2021;22(4):bbaa344.
link1
[31]
Jeon J, Nim S, Teyra J, Datti A, Wrana JL, Sidhu SS, et al. A systematic approach to identify novel cancer drug targets using machine learning, inhibitor design and high-throughput screening. Genome Med 2014;6(7):57.
link1
[32]
Riniker S, Wang Y, Jenkins JL, Landrum GA. Using information from historical high-throughput screens to predict active compounds. J Chem Inf Model 2014;54(7):1880‒91.
link1
[33]
Basile AO, Yahi A, Tatonetti NP. Artificial intelligence for drug toxicity and safety. Trends Pharmacol Sci 2019;40(9):624‒35.
link1
[34]
Cruz Rivera S, Liu X, Chan AW, Denniston AK, Calvert MJ; The SPIRIT-AI and CONSORT-AI Working Group. Guidelines for clinical trial protocols for interventions involving artificial intelligence: the SPIRIT-AI extension. Lancet Digit Health 2020;2(10):e549‒60.
[35]
Steiner S, Wolf J, Glatzel S, Andreou A, Granda JM, Keenan G, et al. Organic synthesis in a modular robotic system driven by a chemical programming language. Science 2019;363(6423):eaav2211.
link1
[36]
Hamet P, Tremblay J. Artificial intelligence in medicine. Metabolism 2017;69(Suppl):S36‒40.
link1
[37]
Zhou Y, Zhang Y, Lian X, Li F, Wang C, Zhu F, et al. Therapeutic target database update 2022: facilitating drug discovery with enriched comparative data of targeted agents. Nucleic Acids Res 2022;50(D1):D1398‒407.
link1
[38]
Amahong K, Zhang W, Zhou Y, Zhang S, Yin J, Li F, et al. CovInter: interaction data between coronavirus RNAs and host proteins. Nucleic Acids Res 2022; 51(D1):D546‒56.
link1
[39]
Liu S, Chen L, Zhang Y, Zhou Y, He Y, Chen Z, et al. M6AREG: m6A-centered regulation of disease development and drug response. Nucleic Acids Res 2022;51(D1):D1333‒44.
link1
[40]
Sun X, Zhang Y, Li H, Zhou Y, Shi S, Chen Z, et al. DRESIS: the first comprehensive landscape of drug resistance information. Nucleic Acids Res 2022;51(D1):D1263‒75.
link1
[41]
Wang X, Li F, Qiu W, Xu B, Li Y, Lian X, et al. SYNBIP: synthetic binding proteins for research, diagnosis and therapy. Nucleic Acids Res 2022;50(D1): D560‒70.
link1
[42]
Zhang S, Sun X, Mou M, Amahong K, Sun H, Zhang W, et al. REGLIV: molecular regulation data of diverse living systems facilitating current multiomics research. Comput Biol Med 2022;148(1):105825.
link1
[43]
Burley SK, Bhikadiya C, Bi C, Bittrich S, Chen L, Crichlow GV, et al. RCSB Protein Data Bank: powerful new tools for exploring 3D structures of biological macromolecules for basic and applied research and education in fundamental biology, biomedicine, biotechnology, bioengineering and energy sciences. Nucleic Acids Res 2021;49(D1):D437‒51.
link1
[44]
Perez-Riverol Y, Bai J, Bandla C, García-Seisdedos D, Hewapathirana S, Kamatchinathan S, et al. The PRIDE database resources in 2022: a hub for mass spectrometry-based proteomics evidences. Nucleic Acids Res 2022; 50(D1):D543‒52.
link1
[45]
Blum M, Chang HY, Chuguransky S, Grego T, Kandasaamy S, Mitchell A, et al. The InterPro protein families and domains database: 20 years on. Nucleic Acids Res 2021;49(D1):D344‒54.
link1
[46]
Yin J, Sun W, Li F, Hong J, Li X, Zhou Y, et al. VARIDT 1.0: variability of drug transporter database. Nucleic Acids Res 2020;48(D1):D1042‒50.
link1
[47]
Fu T, Li F, Zhang Y, Yin J, Qiu W, Li X, et al. VARIDT 2.0: structural variability of drug transporter. Nucleic Acids Res 2022;50(D1):D1417‒31.
link1
[48]
Cunningham F, Allen JE, Allen J, Alvarez-Jarreta J, Amode MR, Armean IM, et al. Ensembl 2022. Nucleic Acids Res 2022;50(D1):D988‒95.
link1
[49]
Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, et al. The human genome browser at UCSC. Genome Res 2002;12(6):996‒1006.
link1
[50]
Barrett T, Wilhite SE, Ledoux P, Evangelista C, Kim IF, Tomashevsky M, et al. NCBI GEO: archive for functional genomics data sets—update. Nucleic Acids Res 2013;41(D1):D991‒5.
link1
[51]
Sayers EW, Cavanaugh M, Clark K, Pruitt KD, Schoch CL, Sherry ST, et al. GenBank. Nucleic Acids Res 2022;50(D1):D161‒4.
link1
[52]
Li W, O’Neill KR, Haft DH, DiCuccio M, Chetvernin V, Badretdin A, et al. RefSeq: expanding the prokaryotic genome annotation pipeline reach with protein family model curation. Nucleic Acids Res 2021;49(D1):D1020‒8.
link1
[53]
Papatheodorou I, Moreno P, Manning J, Fuentes AM, George N, Fexova S, et al. Expression Atlas update: from tissues to single cells. Nucleic Acids Res 2020;48(D1):D77‒83.
[54]
Mendez D, Gaulton A, Bento AP, Chambers J, De Veij M, Félix E, et al. ChEMBL: towards direct deposition of bioassay data. Nucleic Acids Res 2019;47(D1): D930‒40.
link1
[55]
Wishart DS, Feunang YD, Guo AC, Lo EJ, Marcu A, Grant JR, et al. DrugBank 5.0: a major update to the DrugBank database for 2018. Nucleic Acids Res 2018;46 (D1):D1074‒82.
link1
[56]
Li F, Yin J, Lu M, Mou M, Li Z, Zeng Z, et al. DrugMAP: molecular atlas and pharma-information of all drugs. Nucleic Acids Res 2022;51(D1):D1288‒99.
link1
[57]
Tang J, Tanoli ZU, Ravikumar B, Alam Z, Rebane A, Vähä-Koskela M, et al. Drug target commons: a community effort to build a consensus knowledge base for drug‒target interactions. Cell Chem Biol 2018;25(2):224‒9.
link1
[58]
Sheils TK, Mathias SL, Kelleher KJ, Siramshetty VB, Nguyen DT, Bologa CG, et al. TCRD and Pharos 2021: mining the human proteome for disease biology. Nucleic Acids Res 2021;49(D1):D1334‒46.
link1
[59]
Hutter C, Zenklusen JC. The Cancer Genome Atlas: creating lasting value beyond its data. Cell 2018;173(2):283‒5.
link1
[60]
Piñero J, Saüch J, Sanz F, Furlong LI. The DisGeNET Cytoscape App: exploring and visualizing disease genomics data. Comput Struct Biotechnol J 2021;19(1):2960‒7.
link1
[61]
Landrum MJ, Chitipiralla S, Brown GR, Chen C, Gu B, Hart J, et al. ClinVar: improvements to accessing data. Nucleic Acids Res 2020;48(D1):D835‒44.
link1
[62]
Amberger JS, Bocchini CA, Scott AF, OMIMHamosh A..org: leveraging knowledge across phenotype-gene relationships. Nucleic Acids Res 2019;47(D1):D1038‒43.
link1
[63]
Hashimoto DA, Witkowski E, Gao L, Meireles O, Rosman G. Artificial intelligence in anesthesiology: current techniques, clinical applications, and limitations. Anesthesiology 2020;132(2):379‒94.
link1
[64]
Cheng W, Ng CA. Using machine learning to classify bioactivity for 3486 perand polyfluoroalkyl substances (PFASs) from the OECD list. Environ Sci Technol 2019;53(23):13970‒80.
link1
[65]
Hong J, Luo Y, Mou M, Fu J, Zhang Y, Xue W, et al. Convolutional neural network-based annotation of bacterial type IV secretion system effectors with enhanced accuracy and reduced false discovery. Brief Bioinform 2020;21(5):1825‒36.
link1
[66]
Rifaioglu AS, Atas H, Martin MJ, Cetin-Atalay R, Atalay V, Doğan T. Recent applications of deep learning and machine intelligence on in silico drug discovery: methods, tools and databases. Brief Bioinform 2019;20(5):1878‒912.
link1
[67]
Kulmanov M, Hoehndorf R. DeepGOPlus: improved protein function prediction from sequence. Bioinformatics 2020;36(2):422‒9.
link1
[68]
Kulmanov M, Khan MA, Hoehndorf R. DeepGO: predicting protein functions from sequence and interactions using a deep ontology-aware classifier. Bioinformatics 2018;34(4):660‒8.
link1
[69]
Gligorijevic´ V, Renfrew PD, Kosciolek T, Leman JK, Berenberg D, Vatanen T, et al. Structure-based protein function prediction using graph convolutional networks. Nat Commun 2021;12(1):3168.
link1
[70]
Xia W, Zheng L, Fang J, Li F, Zhou Y, Zeng Z, et al. PFmulDL: a novel strategy enabling multi-class and multi-label protein function annotation by integrating diverse deep learning methods. Comput Biol Med 2022;145(1):105465.
link1
[71]
Carracedo-Reboredo P, Liñares-Blanco J, Rodríguez-Fernández N, Cedrón F, Novoa FJ, Carballal A, et al. A review on machine learning approaches and trends in drug discovery. Comput Struct Biotechnol J 2021;19(1):4538‒58.
link1
[72]
Ubels J, Schaefers T, Punt C, Guchelaar HJ, de Ridder J. RAINFOREST: a random forest approach to predict treatment benefit in data from (failed) clinical drug trials. Bioinformatics 2020;36(Suppl 2):i601‒9.
link1
[73]
Yang C, Zhang Y. Delta machine learning to improve scoring-rankingscreening performances of protein‒ligand scoring functions. J Chem Inf Model 2022;62(11):2696‒712.
link1
[74]
Heikamp K, Bajorath J. Support vector machines for drug discovery. Expert Opin Drug Discov 2014;9(1):93‒104.
link1
[75]
Zhang S, Li X, Zong M, Zhu X, Wang R. Efficient kNN classification with different numbers of nearest neighbors. IEEE Trans Neural Netw Learn Syst 2018;29(5):1774‒85.
link1
[76]
Liu B, He H, Luo H, Zhang T, Jiang J. Artificial intelligence and big data facilitated targeted drug discovery. Stroke Vasc Neurol 2019;4(4):206‒13.
link1
[77]
Cirillo D, Valencia A. Big data analytics for personalized medicine. Curr Opin Biotechnol 2019;58(1):161‒7.
link1
[78]
Ma J, Sheridan RP, Liaw A, Dahl GE, Svetnik V. Deep neural nets as a method for quantitative structure‒activity relationships. J Chem Inf Model 2015;55(2):263‒74.
link1
[79]
Shin HC, Roth HR, Gao M, Lu L, Xu Z, Nogues I, et al. Deep convolutional neural networks for computer-aided detection: CNN architectures, dataset characteristics and transfer learning. IEEE Trans Med Imaging 2016;35(5):1285‒98.
link1
[80]
Hou BJ, Zhou ZH. Learning with interpretable structure from gated RNN. IEEE Trans Neural Netw Learn Syst 2020;31(7):2267‒79.
[81]
Zhang Z, Chen L, Zhong F, Wang D, Jiang J, Zhang S, et al. Graph neural network approaches for drug‒target interactions. Curr Opin Struct Biol 2022;73(1):102327.
link1
[82]
Zhang H, Wang Y, Pan Z, Sun X, Mou M, Zhang B, et al. ncRNAInter: a novel strategy based on graph neural network to discover interactions between lncRNA and miRNA. Brief Bioinform 2022;23(6):bbac411.
link1
[83]
Sun C, Cao Y, Wei JM, Liu J. Autoencoder-based drug‒target interaction prediction by preserving the consistency of chemical properties and functions of drugs. Bioinformatics 2021;37(20):3618‒25.
link1
[84]
Yi X, Walia E, Babyn P. Generative adversarial network in medical imaging: a review. Med Image Anal 2019;58(1):101552.
link1
[85]
Zhou X, Shen F, Liu L, Liu W, Nie L, Yang Y, et al. Graph convolutional network hashing. IEEE Trans Cybern 2020;50(4):1460‒72.
link1
[86]
Handelman GS, Kok HK, Chandra RV, Razavi AH, Huang S, Brooks M, et al. Peering into the black box of artificial intelligence: evaluation metrics of machine learning methods. AJR Am J Roentgenol 2019;212(1):38‒43.
link1
[87]
Richards BA, Frankland PW. The persistence and transience of memory. Neuron 2017;94(6):1071‒84.
link1
[88]
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Commun ACM 2017;60(6):84‒90.
link1
[89]
Srivastava N, Hinton G, Krizhevsky A, Sutskever I, Salakhutdinov R. Dropout: a simple way to prevent neural networks from overfitting. J Mach Learn Res 2014;15(1):1929‒58.
[90]
Sun J, Chen X, Zhang Z, Lai S, Zhao B, Liu H, et al. Forecasting the long-term trend of COVID-19 epidemic using a dynamic model. Sci Rep 2020;10(1):21122.
link1
[91]
Jiao Y, Du P. Performance measures in evaluating machine learning based bioinformatics predictors for classifications. Quant Biol 2016;4(4):320‒30.
link1
[92]
Xue L, Bajorath J. Molecular descriptors in chemoinformatics, computational combinatorial chemistry, and virtual screening. Comb Chem High Throughput Screen 2000;3(5):363‒72.
link1
[93]
Wenzel J, Matter H, Schmidt F. Predictive multitask deep neural network models for ADME-Tox properties: learning from large data sets. J Chem Inf Model 2019;59(3):1253‒68.
link1
[94]
Goh GB, Siegel C, Vishnu A, Hodas N. Using rule-based labels for weak supervised learning: a ChemNet for transferable chemical property prediction. In: Proceedings of the 24th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining; 2018 Aug 19‒23; London, UK. New York City: Association for Computing Machinery; 2018. p. 302‒10.
link1
[95]
Popova M, Isayev O, Tropsha A. Deep reinforcement learning for de novo drug design. Sci Adv 2018;4(7):eaap7885.
link1
[96]
Karpov P, Godin G, Tetko IV. Transformer-CNN: Swiss knife for QSAR modeling and interpretation. J Cheminform 2020;12(1):17.
link1
[97]
Goh GB, Hodas NO, Siegel C, Vishnu A. SMILES2Vec: an interpretable generalpurpose deep neural network for predicting chemical properties. 2017. arXiv:171202034.
link1
[98]
Xiong Z, Wang D, Liu X, Zhong F, Wan X, Li X, et al. Pushing the boundaries of molecular representation for drug discovery with the graph attention mechanism. J Med Chem 2020;63(16):8749‒60.
link1
[99]
Yang K, Swanson K, Jin W, Coley C, Eiden P, Gao H, et al. Analyzing learned molecular representations for property prediction. J Chem Inf Model 2019;59(8):3370‒88.
link1
[100]
Li K, Xu C, Huang J, Liu W, Zhang L, Wan W, et al. Prediction and identification of the effectors of heterotrimeric G proteins in rice (Oryza sativa L.). Brief Bioinform 2017;18(2):270‒8.
[101]
Wu M, Yang Y, Wang H, Xu Y. A deep learning method to more accurately recall known lysine acetylation sites. BMC Bioinf 2019;20(1):49.
link1
[102]
Li YH, Xu JY, Tao L, Li XF, Li S, Zeng X, et al. SVM-Prot 2016: a web-server for machine learning prediction of protein functional families from sequence irrespective of similarity. PLoS One 2016;11(8):e0155290.
link1
[103]
Zou L, Nan C, Hu F. Accurate prediction of bacterial type IV secreted effectors using amino acid composition and PSSM profiles. Bioinformatics 2013;29(24):3135‒42.
link1
[104]
Petrilli P. Classification of protein sequences by their dipeptide composition. Comput Appl Biosci 1993;9(2):205‒9.
link1
[105]
Seo S, Oh M, Park Y, Kim S. DeepFam: deep learning based alignment-free method for protein family modeling and prediction. Bioinformatics 2018;34(13):i254‒62.
link1
[106]
Wang J, Yang B, Revote J, Leier A, Marquez-Lago TT, Webb G, et al. POSSUM: a bioinformatics toolkit for generating numerical sequence feature descriptors based on PSSM profiles. Bioinformatics 2017;33(17):2756‒8.
link1
[107]
Hong J, Luo Y, Zhang Y, Ying J, Xue W, Xie T, et al. Protein functional annotation of simultaneously improved stability, accuracy and false discovery rate achieved by a sequence-based deep learning. Brief Bioinform 2020;21(4):1437‒47.
link1
[108]
Yu CY, Li XX, Yang H, Li YH, Xue WW, Chen YZ, et al. Assessing the performances of protein function prediction algorithms from the perspectives of identification accuracy and false discovery rate. Int J Mol Sci 2018;19(1):183.
link1
[109]
Chou KC. Using amphiphilic pseudo amino acid composition to predict enzyme subfamily classes. Bioinformatics 2005;21(1):10‒9.
link1
[110]
Chou KC. Prediction of protein cellular attributes using pseudo-amino acid composition. Proteins 2001;43(3):246‒55.
link1
[111]
Mosier PD, Counterman AE, Jurs PC, Clemmer DE. Prediction of peptide ion collision cross sections from topological molecular structure and amino acid parameters. Anal Chem 2002;74(6):1360‒70.
link1
[112]
Ren B. Atomic-level-based AI topological descriptors for structure‒property correlations. J Chem Inf Comput Sci 2003;43(1):161‒9.
link1
[113]
Magnan CN, Baldi P. SSpro/ACCpro 5: almost perfect prediction of protein secondary structure and relative solvent accessibility using profiles, machine learning and structural similarity. Bioinformatics 2014;30(18):2592‒7.
link1
[114]
Strokach A, Becerra D, Corbi-Verge C, Perez-Riba A, Kim PM. Fast and flexible protein design using deep graph neural networks. Cell Syst 2020;11(4):402‒11.
link1
[115]
Ingraham J, Garg V, Barzilay R, Jaakkola T. Generative models for graph-based protein design. Adv Neural Inf Process Syst 2019;32(1):15820‒31.
[116]
Greener JG, Moffat L, Jones DT. Design of metalloproteins and novel protein folds using variational autoencoders. Sci Rep 2018;8(1):16189.
link1
[117]
Karimi M, Zhu S, Cao Y, Shen Y. De novo protein design for novel folds using guided conditional Wasserstein generative adversarial networks. J Chem Inf Model 2020;60(12):5667‒81.
link1
[118]
Ye Q, Hsieh CY, Yang Z, Kang Y, Chen J, Cao D, et al. A unified drug‒target interaction prediction framework based on knowledge graph and recommendation system. Nat Commun 2021;12(1):6775.
link1
[119]
Rives A, Meier J, Sercu T, Goyal S, Lin Z, Liu J, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci USA 2021;118(15):e2016239118.
link1
[120]
Li GB, Yang LL, Wang WJ, Li LL, Yang SY. ID-Score: a new empirical scoring function based on a comprehensive set of descriptors related to protein‒ ligand interactions. J Chem Inf Model 2013;53(3):592‒600.
link1
[121]
Montanari F, Kuhnke L, Ter Laak A, Clevert DA. Modeling physico-chemical ADMET endpoints with multitask graph convolutional networks. Molecules 2019;25(1):44.
link1
[122]
Dara S, Dhamercherla S, Jadav SS, Babu CM, Ahsan MJ. Machine learning in drug discovery: a review. Artif Intell Rev 2022;55(3):1947‒99.
link1
[123]
Olivecrona M, Blaschke T, Engkvist O, Chen H. Molecular de-novo design through deep reinforcement learning. J Cheminform 2017;9(1):48.
link1
[124]
Dean SN, Alvarez JAE, Zabetakis D, Walper SA, Malanoski AP. PepVAE: variational autoencoder framework for antimicrobial peptide generation and activity prediction. Front Microbiol 2021;12(1):725727.
link1
[125]
Xiong G, Wu Z, Yi J, Fu L, Yang Z, Hsieh C, et al. ADMETlab 2.0: an integrated online platform for accurate and comprehensive predictions of ADMET properties. Nucleic Acids Res 2021;49(W1):W5‒14.
link1
[126]
Gaudelet T, Day B, Jamasb AR, Soman J, Regep C, Liu G, et al. Utilizing graph machine learning within drug discovery and development. Brief Bioinform 2021;22(6):bbab159.
link1
[127]
Swinney DC, Anthony J. How were new medicines discovered? Nat Rev Drug Discov 2011;10(7):507‒19.
link1
[128]
Vincent F, Nueda A, Lee J, Schenone M, Prunotto M, Mercola M. Publisher correction: phenotypic drug discovery: recent successes, lessons learned and new directions. Nat Rev Drug Discov 2022;21(7):541.
link1
[129]
Li YH, Li XX, Hong JJ, Wang YX, Fu JB, Yang H, et al. Clinical trials, progressionspeed differentiating features and swiftness rule of the innovative targets of first-in-class drugs. Brief Bioinform 2020;21(2):649‒62.
link1
[130]
Misra BB, Langefeld CD, Olivier M, Cox LA. Integrated omics: tools, advances, and future approaches. J Mol Endocrinol 2019;62(1):21‒45.
link1
[131]
Fu J, Zhang Y, Wang Y, Zhang H, Liu J, Tang J, et al. Optimization of metabolomic data processing using NOREVA. Nat Protoc 2022;17(1):129‒51.
link1
[132]
Tang J, Fu J, Wang Y, Li B, Li Y, Yang Q, et al. ANPELA: analysis and performance assessment of the label-free quantification workflow for metaproteomic studies. Brief Bioinform 2020;21(2):621‒36.
link1
[133]
Li F, Zhou Y, Zhang Y, Yin J, Qiu Y, Gao J, et al. POSREG: proteomic signature discovered by simultaneously optimizing its reproducibility and generalizability. Brief Bioinform 2022;23(2):bbac040.
link1
[134]
Li F, Yin J, Lu M, Yang Q, Zeng Z, Zhang B, et al. ConSIG: consistent discovery of molecular signature from OMIC data. Brief Bioinform 2022;23(4):bbac253.
link1
[135]
Yang Q, Li B, Wang P, Xie J, Feng Y, Liu Z, et al. LargeMetabo: an out-of-the-box tool for processing and analyzing large-scale metabolomic data. Brief Bioinform 2022;23(6):bbac455.
link1
[136]
Mou M, Pan Z, Lu M, Sun H, Wang Y, Luo Y, et al. Application of machine learning in spatial proteomics. J Chem Inf Model 2022;62(23):5875‒95.
link1
[137]
Fu J, Yang Q, Luo Y, Zhang S, Tang J, Zhang Y, et al. Label-free proteome quantification and evaluation. Brief Bioinform 2022;24(1):bbac477.
link1
[138]
Yang Q, Wang Y, Zhang Y, Li F, Xia W, Zhou Y, et al. NOREVA: enhanced normalization and evaluation of time-course and multi-class metabolomic data. Nucleic Acids Res 2020;48(W1):W436‒48.
link1
[139]
Zhang S, Amahong K, Zhang C, Li F, Gao J, Qiu Y, et al. RNA‒RNA interactions between SARS-CoV-2 and host benefit viral development and evolution during COVID-19 infection. Brief Bioinform 2022;23(1):bbab397.
link1
[140]
Yang Q, Li B, Tang J, Cui X, Wang Y, Li X, et al. Consistent gene signature of schizophrenia identified by a novel feature selection strategy from comprehensive sets of transcriptomic data. Brief Bioinform 2020;21(3):1058‒68.
link1
[141]
Zhang S, Amahong K, Sun X, Lian X, Liu J, Sun H, et al. The miRNA: a small but powerful RNA for COVID-19. Brief Bioinform 2021;22(2):1137‒49.
link1
[142]
Reel PS, Reel S, Pearson E, Trucco E, Jefferson E. Using machine learning approaches for multi-omics data analysis: a review. Biotechnol Adv 2021;49(1):107739.
link1
[143]
The Cancer Genome Atlas Research Network. Comprehensive genomic characterization defines human glioblastoma genes and core pathways. Nature 2008;455(7216):1061‒8. Corrected in: Nature 2013;494(7438):506.
link1
[144]
Subramanian A, Narayan R, Corsello SM, Peck DD, Natoli TE, Lu X, et al. A next generation connectivity map: L1000 platform and the first 1,000,000 profiles. Cell 2017;171(6):1437‒52.
[145]
Uhlén M, Fagerberg L, Hallström BM, Lindskog C, Oksvold P, Mardinoglu A, et al. Tissue-based map of the human proteome. Science 2015;347(6220):1260419.
link1
[146]
Kim MS, Pinto SM, Getnet D, Nirujogi RS, Manda SS, Chaerkady R, et al. A draft map of the human proteome. Nature 2014;509(7502):575‒81.
link1
[147]
Wishart DS, Guo A, Oler E, Wang F, Anjum A, Peters H, et al. HMDB 5.0: the human metabolome database for 2022. Nucleic Acids Res 2022;50(D1): D622‒31.
link1
[148]
Smith CA, O’Maille G, Want EJ, Qin C, Trauger SA, Brandon TR, et al. METLIN: a metabolite mass spectral database. Ther Drug Monit 2005;27(6):747‒51.
link1
[149]
Kanehisa M, Furumichi M, Tanabe M, Sato Y, Morishima K. KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res 2017;45(D1):D353‒61.
link1
[150]
Caspi R, Billington R, Keseler IM, Kothari A, Krummenacker M, Midford PE, et al. The MetaCyc database of metabolic pathways and enzymes—a 2019 update. Nucleic Acids Res 2020;48(D1):D445‒53.
link1
[151]
Gillespie M, Jassal B, Stephan R, Milacic M, Rothfels K, Senff-Ribeiro A, et al. The Reactome pathway knowledgebase 2022. Nucleic Acids Res 2022;50(D1):D687‒92.
link1
[152]
Zhang Y, Tseng JT, Lien IC, Li F, Wu W, Li H. mRNAsi index: machine learning in mining lung adenocarcinoma stem cell biomarkers. Genes 2020;11(3):257.
link1
[153]
Duda M, Zhang H, Li HD, Wall DP, Burmeister M, Guan Y. Brain-specific functional relationship networks inform autism spectrum disorder gene prediction. Transl Psychiatry 2018;8(1):56.
link1
[154]
Liu TP, Hsieh YY, Chou CJ, Yang PM. Systematic polypharmacology and drug repurposing via an integrated L1000-based Connectivity Map database mining. R Soc Open Sci 2018;5(11):181321.
link1
[155]
Gao Y, Kim S, Lee YI, Lee J. Cellular stress-modulating drugs can potentially be identified by in silico screening with Connectivity Map (CMap). Int J Mol Sci 2019;20(22):5601.
link1
[156]
Liu X, Ouyang S, Yu B, Liu Y, Huang K, Gong J, et al. PharmMapper server: a web server for potential drug target identification using pharmacophore mapping approach. Nucleic Acids Res 2010;38(Suppl 2):W609‒14.
link1
[157]
Wang X, Shen Y, Wang S, Li S, Zhang W, Liu X, et al. PharmMapper 2017 update: a web server for potential drug target identification with a comprehensive target pharmacophore database. Nucleic Acids Res 2017;45(W1):W356‒60.
link1
[158]
Wang X, Pan C, Gong J, Liu X, Li H. Enhancing the enrichment of pharmacophore-based target prediction for the polypharmacological profiles of drugs. J Chem Inf Model 2016;56(6):1175‒83.
link1
[159]
Gong J, Cai C, Liu X, Ku X, Jiang H, Gao D, et al. ChemMapper: a versatile web server for exploring pharmacology and chemical structure association based on molecular 3D similarity method. Bioinformatics 2013;29(14):1827‒9.
link1
[160]
Wang X, Chen H, Yang F, Gong J, Li S, Pei J, et al. iDrug: a web-accessible and interactive drug discovery and design platform. J Cheminform 2014;6(1):28.
link1
[161]
Noh H, Gunawan R. Inferring gene targets of drugs and chemical compounds from gene expression profiles. Bioinformatics 2016;32(14):2120‒7.
link1
[162]
Zhu J, Wang J, Wang X, Gao M, Guo B, Gao M, et al. Prediction of drug efficacy from transcriptional profiles with deep learning. Nat Biotechnol 2021;39(11):1444‒52.
link1
[163]
Woo JH, Shimoni Y, Yang WS, Subramaniam P, Iyer A, Nicoletti P, et al. Elucidating compound mechanism of action by network perturbation analysis. Cell 2015;162(2):441‒51.
link1
[164]
Li B, Tang J, Yang Q, Li S, Cui X, Li Y, et al. NOREVA: normalization and evaluation of MS-based metabolomics data. Nucleic Acids Res 2017;45(W1): W162‒70.
link1
[165]
The Cancer Genome Atlas Research Network. Integrated genomic analyses of ovarian carcinoma. Nature 2011;474(7353):609‒15. Erratum in: Nature 2012;490(7419):292.
link1
[166]
Masica DL, Karchin R. Correlation of somatic mutation and expression identifies genes important in human glioblastoma progression and survival. Cancer Res 2011;71(13):4550‒61.
link1
[167]
Fang J, Zhang P, Wang Q, Chiang CW, Zhou Y, Hou Y, et al. Artificial intelligence framework identifies candidate targets for drug repurposing in Alzheimer’s disease. Alzheimers Res Ther 2022;14(1):7.
link1
[168]
Pabon NA, Xia Y, Estabrooks SK, Ye Z, Herbrand AK, Süß E, et al. Predicting protein targets for drug-like compounds using transcriptomics. PLoS Comput Biol 2018;14(12):e1006651.
link1
[169]
Zhong F, Wu X, Yang R, Li X, Wang D, Fu Z, et al. Drug target inference by mining transcriptional data using a novel graph convolutional network framework. Protein Cell 2022;13(4):281‒301.
link1
[170]
Jaganathan K, Kyriazopoulou Panagiotopoulou S, McRae JF, Darbandi SF, Knowles D, Li YI, et al. Predicting splicing from primary sequence with deep learning. Cell 2019;176(3):535‒48.
link1
[171]
Lopez R, Regier J, Cole MB, Jordan MI, Yosef N. Deep generative modeling for single-cell transcriptomics. Nat Methods 2018;15(12):1053‒8.
link1
[172]
Liu F, Li H, Ren C, Bo X, Shu W. PEDLA: predicting enhancers with a deep learning-based algorithmic framework. Sci Rep 2016;6(1):28517.
link1
[173]
Downes DJ, Cross AR, Hua P, Roberts N, Schwessinger R, Cutler AJ, et al.; COMBAT Consortium. Identification of LZTFL1 as a candidate effector gene at a COVID-19 risk locus. Nat Genet 2021;53(11):1606‒15.
link1
[174]
Barretina J, Caponigro G, Stransky N, Venkatesan K, Margolin AA, Kim S, et al. The cancer cell line encyclopedia enables predictive modelling of anticancer drug sensitivity. Nature 2012;483(7391):603‒7.
[175]
Dry JR, Pavey S, Pratilas CA, Harbron C, Runswick S, Hodgson D, et al. Transcriptional pathway signatures predict MEK addiction and response to selumetinib (AZD6244). Cancer Res 2010;70(6):2264‒73.
link1
[176]
Sharifi-Noghabi H, Zolotareva O, Collins CC, Ester M. MOLI: multi-omics late integration with deep neural networks for drug response prediction. Bioinformatics 2019;35(14):i501‒9.
link1
[177]
Iorio F, Knijnenburg TA, Vis DJ, Bignell GR, Menden MP, Schubert M, et al. A landscape of pharmacogenomic interactions in cancer. Cell 2016;166(3):740‒54.
link1
[178]
Peng W, Chen T, Dai W. Predicting drug response based on multi-omics fusion and graph convolution. IEEE J Biomed Health Inform 2022;26 (3):1384‒93.
link1
[179]
Wang Y, Yang Y, Chen S, Wang J. DeepDRK: a deep learning framework for drug repurposing through kernel-based multi-omics integration. Brief Bioinform 2021;22(5):bbab048.
link1
[180]
Novac N. Challenges and opportunities of drug repositioning. Trends Pharmacol Sci 2013;34(5):267‒72.
link1
[181]
Keiser MJ, Setola V, Irwin JJ, Laggner C, Abbas AI, Hufeisen SJ, et al. Predicting new molecular targets for known drugs. Nature 2009;462(7270):175‒81.
link1
[182]
Davis AP, Grondin CJ, Johnson RJ, Sciaky D, Wiegers J, Wiegers TC, et al. Comparative Toxicogenomics Database (CTD): update 2021. Nucleic Acids Res 2021;49(D1):D1138‒43.
link1
[183]
Harding SD, Armstrong JF, Faccenda E, Southan C, Alexander SPH, Davenport AP, et al. The IUPHAR/BPS guide to PHARMACOLOGY in 2022: curating pharmacology for COVID-19, malaria and antibacterials. Nucleic Acids Res 2022;50(D1):D1282‒94.
link1
[184]
Avram S, Bologa CG, Holmes J, Bocci G, Wilson TB, Nguyen DT, et al. DrugCentral 2021 supports drug discovery and repositioning. Nucleic Acids Res 2021;49(D1):D1160‒9.
link1
[185]
Urán Landaburu L, Berenstein AJ, Videla S, Maru P, Shanmugam D, Chernomoretz A, et al. TDR Targets 6: driving drug discovery for human pathogens through intensive chemogenomic data integration. Nucleic Acids Res 2020;48(D1):D992‒1005.
[186]
Chen TF, Chang YC, Hsiao Y, Lee KH, Hsiao YC, Lin YH, et al. DockCoV2: a drug database against SARS-CoV-2. Nucleic Acids Res 2021;49(D1):D1152‒9.
link1
[187]
Kanehisa M, Furumichi M, Sato Y, Ishiguro-Watanabe M, Tanabe M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Res 2021;49(D1): D545‒51.
link1
[188]
Wang C, Hu G, Wang K, Brylinski M, Xie L, Kurgan L. PDID: database of molecular-level putative protein‒drug interactions in the structural human proteome. Bioinformatics 2016;32(4):579‒86.
link1
[189]
Kuhn M, Letunic I, Jensen LJ, Bork P. The SIDER database of drugs and side effects. Nucleic Acids Res 2016;44(D1):D1075‒9.
link1
[190]
Ochoa D, Hercules A, Carmona M, Suveges D, Gonzalez-Uriarte A, Malangone C, et al. Open Targets Platform: supporting systematic drug‒target identification and prioritisation. Nucleic Acids Res 2021;49(D1):D1302‒10.
link1
[191]
Gao Z, Li H, Zhang H, Liu X, Kang L, Luo X, et al. PDTD: a web-accessible protein database for drug target identification. BMC Bioinf 2008;9(1):104.
link1
[192]
RDKit: open-source cheminformatics software [Internet]. Basel: T5 Informatics GmbH; [cited 2023 Feb 9]. Available from: https://www.rdkit. org/.
link1
[193]
O’Boyle NM, Banck M, James CA, Morley C, Vandermeersch T, Hutchison GR. Open Babel: an open chemical toolbox. J Cheminform 2011;3(1):33.
link1
[194]
Daylight Toolkit: C-language interface for SMILESTM, SMARTS®, and SMIRKS® [Internet]. Laguna Niguel: Daylight Chemical Information Systems, Inc.; [cited 2023 Feb 9]. Available from: https://www.daylight.com/products/toolkit.html.
link1
[195]
Steinbeck C, Han Y, Kuhn S, Horlacher O, Luttmann E, Willighagen E. The Chemistry Development Kit (CDK): an open-source Java library for chemoand bioinformatics. J Chem Inf Comput Sci 2003;43(2):493‒500.
link1
[196]
ToolkitsOpenEye 2022.2.2 [Internet]. Santa Fe: OpenEye Scientific Software, Inc.; [cited 2023 Feb 9]. Available from: https://docs.eyesopen.com/toolkits/ python/index.html.
link1
[197]
Cao Y, Charisi A, Cheng LC, Jiang T, Girke T. ChemmineR: a compound mining framework for R. Bioinformatics 2008;24(15):1733‒4.
link1
[198]
Indigo Toolkit [Internet]. Newtown: EPAM System, Inc.; [cited 2023 Feb 9]. Available from: https://lifescience.opensource.epam.com/indigo/.
link1
[199]
Liu X, Jiang H, Li H. SHAFTS: a hybrid approach for 3D molecular similarity calculation. 1. Method and assessment of virtual screening. J Chem Inf Model 2011;51(9):2372‒85.
link1
[200]
Lu W, Liu X, Cao X, Xue M, Liu K, Zhao Z, et al. SHAFTS: a hybrid approach for 3D molecular similarity calculation. 2. Prospective case study in the discovery of diverse p90 ribosomal S6 protein kinase 2 inhibitors to suppress cell migration. J Med Chem 2011;54(10):3564‒74.
link1
[201]
He G, Song Y, Wei W, Wang X, Lu X, Li H. eSHAFTS: integrated and graphical drug design software based on 3D molecular similarity. J Comput Chem 2019;40(6):826‒38.
link1
[202]
Zhang P, Tao L, Zeng X, Qin C, Chen S, Zhu F, et al. A protein network descriptor server and its use in studying protein, disease, metabolic and drug targeted networks. Brief Bioinform 2017;18(6):1057‒70.
[203]
Boratyn GM, Camacho C, Cooper PS, Coulouris G, Fong A, Ma N, et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Res 2013;41(W1):W29‒33.
link1
[204]
Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res 1994;22(22):4673‒80.
link1
[205]
Holm L, Laakso LM. Dali server update. Nucleic Acids Res 2016;44(W1): W351‒5.
link1
[206]
Shatsky M, Nussinov R, Wolfson HJ. A method for simultaneous alignment of multiple protein structures. Proteins 2004;56(1):143‒56.
link1
[207]
Zhang Y, Skolnick J. TM-align: a protein structure alignment algorithm based on the TM-score. Nucleic Acids Res 2005;33(7):2302‒9.
link1
[208]
Li S, Cai C, Gong J, Liu X, Li H. A fast protein binding site comparison algorithm for proteome-wide protein function prediction and drug repurposing. Proteins 2021;89(11):1541‒56.
link1
[209]
Prlić A, Bliven S, Rose PW, Bluhm WF, Bizon C, Godzik A, et al. Pre-calculated protein structure alignments at the RCSB PDB website. Bioinformatics 2010;26(23):2983‒5.
link1
[210]
Shulman-Peleg A, Nussinov R, Wolfson HJ. Recognition of functional sites in protein structures. J Mol Biol 2004;339(3):607‒33.
link1
[211]
Gao M, Skolnick J. APoc: large-scale identification of similar protein pockets. Bioinformatics 2013;29(5):597‒604.
link1
[212]
Brylinski M. eMatchSite: sequence order-independent structure alignments of ligand binding pockets in protein models. PLoS Comput Biol 2014;10(9): e1003829.
link1
[213]
Björkholm P, Daniluk P, Kryshtafovych A, Fidelis K, Andersson R, Hvidsten TR. Using multi-data hidden Markov models trained on local neighborhoods of protein structure to predict residue‒residue contacts. Bioinformatics 2009;25(10):1264‒70.
link1
[214]
McGuffin LJ, Bryson K, Jones DT. The PSIPRED protein structure prediction server. Bioinformatics 2000;16(4):404‒5.
link1
[215]
Nayal M, Honig B. On the nature of cavities on protein surfaces: application to the identification of drug-binding sites. Proteins 2006;63(4):892‒906.
link1
[216]
Cao DS, Liu S, Xu QS, Lu HM, Huang JH, Hu QN, et al. Large-scale prediction of drug‒target interactions using protein sequences and drug topological structures. Anal Chim Acta 2012;752(1):1‒10.
link1
[217]
Öztürk H, Özgür A, Ozkirimli E. DeepDTA: deep drug‒target binding affinity prediction. Bioinformatics 2018;34(17):i821‒9.
link1
[218]
Rayhan F, Ahmed S, Shatabda S, Farid DM, Mousavian Z, Dehzangi A, et al. iDTI-ESBoost: identification of drug target interaction using evolutionary and structural features with boosting. Sci Rep 2017;7(1):17731.
link1
[219]
Huang K, Fu T, Glass LM, Zitnik M, Xiao C, Sun J. DeepPurpose: a deep learning library for drug‒target interaction prediction. Bioinformatics 2021;36(22‒23):5545‒7.
[220]
Bleakley K, Yamanishi Y. Supervised prediction of drug‒target interactions using bipartite local models. Bioinformatics 2009;25(18):2397‒403.
link1
[221]
Yamanishi Y, Araki M, Gutteridge A, Honda W, Kanehisa M. Prediction of drug‒target interaction networks from the integration of chemical and genomic spaces. Bioinformatics 2008;24(13):i232‒40.
link1
[222]
Yıldırım MA, Goh KI, Cusick ME, Barabási AL, Vidal M. Drug‒target network. Nat Biotechnol 2007;25(10):1119‒26.
link1
[223]
Luo Y, Zhao X, Zhou J, Yang J, Zhang Y, Kuang W, et al. A network integration approach for drug‒target interaction prediction and computational drug repositioning from heterogeneous information. Nat Commun 2017;8(1):573.
link1
[224]
Zeng X, Zhu S, Lu W, Liu Z, Huang J, Zhou Y, et al. Target identification among known drugs by deep learning from heterogeneous networks. Chem Sci 2020;11(7):1775‒97.
link1
[225]
Mohamed SK, Nounu A, Novácˇek V. Biological applications of knowledge graph embedding models. Brief Bioinform 2021;22(2):1679‒93.
link1
[226]
Perlman L, Gottlieb A, Atias N, Ruppin E, Sharan R. Combining drug and gene similarity measures for drug‒target elucidation. J Comput Biol 2011;18(2):133‒45.
link1
[227]
Cobanoglu MC, Liu C, Hu F, Oltvai ZN, Bahar I. Predicting drug‒target interactions using probabilistic matrix factorization. J Chem Inf Model 2013;53(12):3399‒409.
link1
[228]
Sydow D, Burggraaff L, Szengel A, van Vlijmen HWT, IJzerman AP, van Westen GJP, et al. Advances and challenges in computational target prediction. J Chem Inf Model 2019;59(5):1728‒42.
link1
[229]
Bagherian M, Sabeti E, Wang K, Sartor MA, Nikolovska-Coleska Z, Najarian K. Machine learning approaches and databases for prediction of drug‒target interaction: a survey paper. Brief Bioinform 2021;22(1):247‒69.
link1
[230]
Zhang X, Li L, Ng MK, Zhang S. Drug‒target interaction prediction by integrating multiview network data. Comput Biol Chem 2017;69(1):185‒93.
link1
[231]
Zhang W, Chen Y, Li D. Drug‒target interaction prediction through label propagation with linear neighborhood information. Molecules 2017;22(12):2056.
link1
[232]
van Laarhoven T, Marchiori E. Predicting drug‒target interactions for new drug compounds using a weighted nearest neighbor profile. PLoS One 2013;8(6):e66952.
link1
[233]
He T, Heidemeyer M, Ban F, Cherkasov A, Ester M. SimBoost: a read-across approach for predicting drug‒target binding affinities using gradient boosting machines. J Cheminform 2017;9(1):24.
link1
[234]
Sharma A, Rani R. BE-DTI’: ensemble framework for drug target interaction prediction using dimensionality reduction and active learning. Comput Methods Programs Biomed 2018;165(1):151‒62.
link1
[235]
Liu Y, Wu M, Miao C, Zhao P, Li XL. Neighborhood regularized logistic matrix factorization for drug‒target interaction prediction. PLoS Comput Biol 2016;12(2):e1004760.
link1
[236]
Bolgár B, Antal P. VB-MK-LMF: fusion of drugs, targets and interactions using variational Bayesian multiple kernel logistic matrix factorization. BMC Bioinf 2017;18(1):440.
link1
[237]
Li L, Cai M. Drug target prediction by multi-view low rank embedding. IEEE/ ACM Trans Comput Biol Bioinform 2019;16(5):1712‒21.
link1
[238]
Cheng F, Liu C, Jiang J, Lu W, Li W, Liu G, et al. Prediction of drug‒target interactions and drug repositioning via network-based inference. PLoS Comput Biol 2012;8(5):e1002503.
link1
[239]
Chen X, Liu MX, Yan GY. Drug‒target interaction prediction by random walk on the heterogeneous network. Mol Biosyst 2012;8(7):1970‒8.
link1
[240]
Chen H, Zhang Z. A semi-supervised method for drug‒target interaction prediction with consistency in networks. PLoS One 2013;8(5):e62975.
link1
[241]
Alaimo S, Pulvirenti A, Giugno R, Ferro A. Drug‒target interaction prediction through domain-tuned network-based inference. Bioinformatics 2013;29(16):2004‒8.
link1
[242]
Mongia A, Majumdar A. Drug‒target interaction prediction using multi graph regularized nuclear norm minimization. PLoS One 2020;15(1):e0226484.
link1
[243]
Wang Y, Zeng J. Predicting drug‒target interactions using restricted Boltzmann machines. Bioinformatics 2013;29(13):i126‒34.
link1
[244]
Shi H, Liu S, Chen J, Li X, Ma Q, Yu B. Predicting drug‒target interactions using Lasso with random forest based on evolutionary information and chemical structure. Genomics 2019;111(6):1839‒52.
link1
[245]
Wen M, Zhang Z, Niu S, Sha H, Yang R, Yun Y, et al. Deep-learning-based drug‒target interaction prediction. J Proteome Res 2017;16(4):1401‒9.
link1
[246]
Lee I, Keum J, Nam H. DeepConv-DTI: prediction of drug‒target interactions via deep learning with convolution on protein sequences. PLoS Comput Biol 2019;15(6):e1007129.
link1
[247]
Xie L, He S, Song X, Bo X, Zhang Z. Deep learning-based transcriptome data classification for drug‒target interaction prediction. BMC Genomics 2018;19(Suppl 7):667.
link1
[248]
Verma J, Khedkar VM, Coutinho EC. 3D-QSAR in drug design—a review. Curr Top Med Chem 2010;10(1):95‒115.
link1
[249]
Jing Y, Bian Y, Hu Z, Wang L, Xie XQ. Deep learning for drug design: an artificial intelligence paradigm for drug discovery in the big data era. AAPS J 2018;20(3):58. Corrected in: AAPS J 2018;20(4):79.
link1
[250]
Hessler G, Baringhaus KH. Artificial intelligence in drug design. Molecules 2018;23(10):2520.
link1
[251]
Burello E, Worth AP. QSAR modeling of nanomaterials. Wiley Interdiscip Rev Nanomed Nanobiotechnol 2011;3(3):298‒306.
link1
[252]
Xue W, Fu T, Deng S, Yang F, Yang J, Zhu F. Molecular mechanism for the allosteric inhibition of the human serotonin transporter by antidepressant escitalopram. ACS Chem Neurosci 2022;13(3):340‒51.
link1
[253]
Ballante F, Kooistra AJ, Kampen S, de Graaf C, Carlsson J. Structure-based virtual screening for ligands of G protein-coupled receptors: what can molecular docking do for you? Pharmacol Rev 2021;73(4):1698‒736.
link1
[254]
Shin WH, Zhu X, Bures MG, Kihara D. Three-dimensional compound comparison methods and their application in drug discovery. Molecules 2015;20(7):12841‒62.
link1
[255]
Ghislat G, Rahman T, Ballester PJ. Recent progress on the prospective application of machine learning to structure-based virtual screening. Curr Opin Chem Biol 2021;65(1):28‒34.
link1
[256]
Liu M, Wang S. MCDOCK: a Monte Carlo simulation approach to the molecular docking problem. J Comput Aided Mol Des 1999;13(5):435‒51.
link1
[257]
Sneha P, George Priya Doss C. Molecular dynamics: new frontier in personalized medicine. Adv Protein Chem Struct Biol 2016;102(1): 181‒224.
[258]
Xue W, Yang F, Wang P, Zheng G, Chen Y, Yao X, et al. What contributes to serotonin‒norepinephrine reuptake inhibitors’ dual-targeting mechanism? The key role of transmembrane domain 6 in human serotonin and norepinephrine transporters revealed by molecular dynamics simulation. ACS Chem Neurosci 2018;9(5):1128‒40.
link1
[259]
Xie QQ, Zhong L, Pan YL, Wang XY, Zhou JP, Di-wu L, et al. Combined SVMbased and docking-based virtual screening for retrieving novel inhibitors of c-Met. Eur J Med Chem 2011;46(9):3675‒80.
link1
[260]
Pereira JC, Caffarena ER, Dos Santos CN. Boosting docking-based virtual screening with deep learning. J Chem Inf Model 2016;56(12):2495‒506.
link1
[261]
Huang N, Shoichet BK, Irwin JJ. Benchmarking sets for molecular docking. J Med Chem 2006;49(23):6789‒801.
link1
[262]
Lang PT, Brozell SR, Mukherjee S, Pettersen EF, Meng EC, Thomas V, et al. DOCK 6: combining techniques to model RNA—small molecule complexes. RNA 2009;15(6):1219‒30.
link1
[263]
Trott O, Olson AJ. AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading. J Comput Chem 2010;31(2):455‒61.
link1
[264]
AbdulHameed MD, Ippolito DL, Wallqvist A. Predicting rat and human pregnane X receptor activators using Bayesian classification models. Chem Res Toxicol 2016;29(10):1729‒40.
link1
[265]
Martin EJ, Polyakov VR, Tian L, Perez RC. Profile-QSAR 2.0: kinase virtual screening accuracy comparable to four-concentration IC50s for realistically novel compounds. J Chem Inf Model 2017;57(8):2077‒88.
[266]
Chen JJF, Visco Jr DP. Developing an in silico pipeline for faster drug candidate discovery: virtual high throughput screening with the signature molecular descriptor using support vector machine models. Chem Eng Sci 2017;159(1):31‒42.
link1
[267]
Myint KZ, Wang L, Tong Q, Xie XQ. Molecular fingerprint-based artificial neural networks QSAR for ligand biological activity predictions. Mol Pharm 2012;9(10):2912‒23.
link1
[268]
Jaén-Oltra J, Salabert-Salvador MT, García-March FJ, Pérez-Giménez F, Tomás-Vert F. Artificial neural network applied to prediction of fluorquinolone antibacterial activity by topological methods. J Med Chem 2000;43(6):1143‒8.
link1
[269]
Lenselink EB, Ten Dijke N, Bongers B, Papadatos G, van Vlijmen HWT, Kowalczyk W, et al. Beyond the hype: deep neural networks outperform established methods using a ChEMBL bioactivity benchmark set. J Cheminform 2017;9(1):45.
link1
[270]
Keiser MJ, Roth BL, Armbruster BN, Ernsberger P, Irwin JJ, Shoichet BK. Relating protein pharmacology by ligand chemistry. Nat Biotechnol 2007;25(2):197‒206.
link1
[271]
Xiao T, Qi X, Chen Y, Jiang Y. Development of ligand-based big data deep neural network models for virtual screening of large compound libraries. Mol Inform 2018;37(11):1800031.
link1
[272]
Fang J, Wang L, Li Y, Lian W, Pang X, Wang H, et al. AlzhCPI: a knowledge base for predicting chemical‒protein interactions towards Alzheimer’s disease. PLoS One 2017;12(5):e0178347.
link1
[273]
Bender A, Mussa HY, Glen RC. Screening for dihydrofolate reductase inhibitors using MOLPRINT 2D, a fast fragment-based method employing the naïve Bayesian classifier: limitations of the descriptor and the importance of balanced chemistry in training and test sets. J Biomol Screen 2005;10(7):658‒66.
link1
[274]
Abdo A, Chen B, Mueller C, Salim N, Willett P. Ligand-based virtual screening using Bayesian networks. J Chem Inf Model 2010;50(6):1012‒20.
link1
[275]
Li Y, Wang L, Liu Z, Li C, Xu J, Gu Q, et al. Predicting selective liver X receptor b agonists using multiple machine learning methods. Mol Biosyst 2015;11(5):1241‒50.
link1
[276]
Fang J, Yang R, Gao L, Zhou D, Yang S, Liu AL, et al. Predictions of BuChE inhibitors using support vector machine and naive Bayesian classification techniques in drug discovery. J Chem Inf Model 2013;53(11):3009‒20.
link1
[277]
Schneider P, Tanrikulu Y, Schneider G. Self-organizing maps in drug discovery: compound library design, scaffold-hopping, repurposing. Curr Med Chem 2009;16(3):258‒66.
link1
[278]
Hristozov D, Oprea TI, Gasteiger J. Ligand-based virtual screening by novelty detection with self-organizing maps. J Chem Inf Model 2007;47(6):2044‒62.
link1
[279]
Reker D, Rodrigues T, Schneider P, Schneider G. Identifying the macromolecular targets of de novo-designed chemical entities through selforganizing map consensus. Proc Natl Acad Sci USA 2014;111(11):4067‒72.
link1
[280]
Stojanović L, Popović M, Tijanić N, Rakočević G, Kalinić M. Improved scaffold hopping in ligand-based virtual screening using neural representation learning. J Chem Inf Model 2020;60(10):4629‒39.
link1
[281]
Kadurin A, Aliper A, Kazennov A, Mamoshina P, Vanhaelen Q, Khrabrov K, et al. The cornucopia of meaningful leads: applying deep adversarial autoencoders for new molecule development in oncology. Oncotarget 2017;8(7):10883‒90.
link1
[282]
Xu Y, Chen P, Lin X, Yao H, Lin K. Discovery of CDK4 inhibitors by convolutional neural networks. Future Med Chem 2019;11(3):165‒77.
link1
[283]
Altae-Tran H, Ramsundar B, Pappu AS, Pande V. Low data drug discovery with one-shot learning. ACS Cent Sci 2017;3(4):283‒93.
link1
[284]
Zhou Z, Kearnes S, Li L, Zare RN, Riley P. Optimization of molecules via deep reinforcement learning. Sci Rep 2019;9(1):10752. Corrected in: Sci Rep 2020;10(1):10478.
link1
[285]
Hartenfeller M, Schneider G. De novo drug design. Methods Mol Biol 2011;672(1):299‒323.
[286]
Segall M. Advances in multiparameter optimization methods for de novo drug design. Expert Opin Drug Discov 2014;9(7):803‒17.
link1
[287]
Schneider G, Fechner U. Computer-based de novo design of drug-like molecules. Nat Rev Drug Discov 2005;4(8):649‒63.
link1
[288]
Sohn JI, Nam JW. The present and future of de novo whole-genome assembly. Brief Bioinform 2018;19(1):23‒40.
[289]
Schneider G, Clark DE. Automated de novo drug design: are we nearly there yet? Angew Chem Int Ed Engl 2019;58(32):10792‒803.
link1
[290]
Xiong J, Xiong Z, Chen K, Jiang H, Zheng M. Graph neural networks for automated de novo drug design. Drug Discov Today 2021;26(6):1382‒93.
link1
[291]
Pereira T, Abbasi M, Ribeiro B, Arrais JP. Diversity oriented deep reinforcement learning for targeted molecule generation. J Cheminform 2021;13(1):21.
link1
[292]
Ståhl N, Falkman G, Karlsson A, Mathiason G, Boström J. Deep reinforcement learning for multiparameter optimization in de novo drug design. J Chem Inf Model 2019;59(7):3166‒76.
[293]
Maziarka Ł, Pocha A, Kaczmarczyk J, Rataj K, Danel T, Warchoł M. Mol- CycleGAN: a generative model for molecular optimization. J Cheminform 2020;12(1):2.
link1
[294]
Sanchez-Lengeling B, Outeiral C, Guimaraes GL, Aspuru-Guzik A. Optimizing distributions over molecular space. An objective-reinforced generative adversarial network for inverse-design chemistry (ORGANIC). ChemRxiv. Cambridge: Cambridge Open Engage; 2017.
[295]
Putin E, Asadulaev A, Ivanenkov Y, Aladinskiy V, Sanchez-Lengeling B, Aspuru-Guzik A, et al. Reinforced adversarial neural computer for de novo molecular design. J Chem Inf Model 2018;58(6):1194‒204.
link1
[296]
Harel S, Radinsky K. Prototype-based compound discovery using deep generative models. Mol Pharm 2018;15(10):4406‒16.
link1
[297]
Wilman W, Wrobel S, Bielska W, Deszynski P, Dudzic P, Jaszczyszyn I, et al. Machine-designed biotherapeutics: opportunities, feasibility and advantages of deep learning in computational antibody discovery. Brief Bioinform 2022;23(4):bbac267.
link1
[298]
Ruffolo JA, Sulam J, Gray JJ. Antibody structure prediction using interpretable deep learning. Patterns 2022;3(2):100406.
link1
[299]
Sivasubramanian A, Sircar A, Chaudhury S, Gray JJ. Toward high-resolution homology modeling of antibody Fv regions and application to antibodyantigen docking. Proteins 2009;74(2):497‒514.
link1
[300]
Schneider C, Buchanan A, Taddese B, Deane CM. DLAB: deep learning methods for structure-based virtual screening of antibodies. Bioinformatics 2022;38(2):377‒83.
link1
[301]
Eguchi RR, Choe CA, Huang PS. Ig-VAE: generative modeling of protein structure by direct 3D coordinate generation. PLoS Comput Biol 2022;18(6): e1010271.
link1
[302]
Raybould MIJ, Marks C, Krawczyk K, Taddese B, Nowak J, Lewis AP, et al. Five computational developability guidelines for therapeutic antibody profiling. Proc Natl Acad Sci USA 2019;116(10):4025‒30.
link1
[303]
Kim JH, Hong HJ. Humanization by CDR grafting and specificity-determining residue grafting. Methods Mol Biol 2012;907(1):237‒45.
[304]
Leem J, Mitchell LS, Farmery JHR, Barton J, Galson JD. Deciphering the language of antibodies using self-supervised learning. Patterns 2022;3(7):100513.
link1
[305]
Olsen TH, Moal IH, Deane CM. AbLang: an antibody language model for completing antibody sequences. Bioinform Adv 2022;2(1):vbac046.
link1
[306]
Fu J, Zhang Y, Liu J, Lian X, Tang J, Zhu F. Pharmacometabonomics: data processing and statistical analysis. Brief Bioinform 2021;22(5): bbab138.
link1
[307]
Meanwell NA. Improving drug candidates by design: a focus on physicochemical properties as a means of improving compound disposition and safety. Chem Res Toxicol 2011;24(9):1420‒56.
link1
[308]
Lipinski CA. Lead- and drug-like compounds: the rule-of-five revolution. Drug Discov Today Technol 2004;1(4):337‒41.
link1
[309]
Zhang MQ, Wilkinson B. Drug discovery beyond the ‘rule-of-five’. Curr Opin Biotechnol 2007;18(6):478‒88.
link1
[310]
Manallack DT, Prankerd RJ, Yuriev E, Oprea TI, Chalmers DK. The significance of acid/base properties in drug discovery. Chem Soc Rev 2013;42(2):485‒96.
link1
[311]
Zhang H, Xiang ML, Ma CY, Huang Q, Li W, Xie Y, et al. Three-class classification models of logS and logP derived by using GA-CG-SVM approach. Mol Divers 2009;13(2):261‒8.
link1
[312]
Jorgensen WL, Duffy EM. Prediction of drug solubility from Monte Carlo simulations. Bioorg Med Chem Lett 2000;10(11):1155‒8.
link1
[313]
Kamlet MJ, Doherty RM, Abboud JL, Abraham MH, Taft RW. Linear solvation energy relationships: 36. molecular properties governing solubilities of organic nonelectrolytes in water. J Pharm Sci 1986;75(4):338‒49.
link1
[314]
Yang X, Wang Y, Byrne R, Schneider G, Yang S. Concepts of artificial intelligence for computer-assisted drug discovery. Chem Rev 2019;119(18):10520‒94.
link1
[315]
Elder D, Holm R. Aqueous solubility: simple predictive methods (in silico, in vitro and bio-relevant approaches). Int J Pharm 2013;453(1):3‒11.
link1
[316]
Hewitt M, Cronin MT, Enoch SJ, Madden JC, Roberts DW, Dearden JC. In silico prediction of aqueous solubility: the solubility challenge. J Chem Inf Model 2009;49(11):2572‒87.
link1
[317]
Francoeur PG, Koes DR. SolTranNet—a machine learning tool for fast aqueous solubility prediction. J Chem Inf Model 2021;61(6):2530‒6.
link1
[318]
Rogers D, Hahn M. Extended-connectivity fingerprints. J Chem Inf Model 2010;50(5):742‒54.
link1
[319]
Shen WX, Zeng X, Zhu F, Wang YL, Qin C, Tan Y, et al. Out-of-the-box deep learning prediction of pharmaceutical properties by broadly learned knowledge-based molecular representations. Nat Mach Intell 2021;3(4):334‒43.
link1
[320]
Yin J, Li F, Zhou Y, Mou M, Lu Y, Chen K, et al. INTEDE: interactome of drugmetabolizing enzymes. Nucleic Acids Res 2021;49(D1):D1233‒43.
link1
[321]
Cheng F, Li W, Liu G, Tang Y. In silico ADMET prediction: recent advances, current challenges and future trends. Curr Top Med Chem 2013;13(11):1273‒89.
link1
[322]
Wang Y, Xing J, Xu Y, Zhou N, Peng J, Xiong Z, et al. In silico ADME/T modelling for rational drug design. Q Rev Biophys 2015;48(4):488‒515.
link1
[323]
Ferreira LLG, Andricopulo AD. ADMET modeling approaches in drug discovery. Drug Discov Today 2019;24(5):1157‒65.
link1
[324]
Tao L, Zhang P, Qin C, Chen SY, Zhang C, Chen Z, et al. Recent progresses in the exploration of machine learning methods as in-silico ADME prediction tools. Adv Drug Deliv Rev 2015;86(1):83‒100.
link1
[325]
Rácz A, Bajusz D, Miranda-Quintana RA, Héberger K. Machine learning models for classification tasks related to drug safety. Mol Divers 2021;25(3):1409‒24.
link1
[326]
Vandenberg JI, Perry MD, Perrin MJ, Mann SA, Ke Y, Hill AP. hERG K+ channels: structure, function, and clinical significance. Physiol Rev 2012;92(3):1393‒478.
link1
[327]
Kaisar MA, Sajja RK, Prasad S, Abhyankar VV, Liles T, Cucullo L. New experimental models of the blood‒brain barrier for CNS drug discovery. Expert Opin Drug Discov 2017;12(1):89‒103.
link1
[328]
Smyth MJ, Krasovskis E, Sutton VR, Johnstone RW. The drug efflux protein, P-glycoprotein, additionally protects drug-resistant tumor cells from multiple forms of caspase-dependent apoptosis. Proc Natl Acad Sci USA 1998;95(12):7024‒9.
link1
[329]
Rácz A, Keser}u GM. Large-scale evaluation of cytochrome P450 2C9 mediated drug interaction potential with machine learning-based consensus modeling. J Comput Aided Mol Des 2020;34(8):831‒9.
link1
[330]
Yang H, Sun L, Li W, Liu G, Tang Y. In silico prediction of chemical toxicity for drug design using machine learning methods and structural alerts. Front Chem 2018;6(1):30.
link1
[331]
Onakpoya IJ, Heneghan CJ, Aronson JK. Post-marketing withdrawal of 462 medicinal products because of adverse drug reactions: a systematic review of the world literature. BMC Med 2016;14(1):10. Corrected in: BMC Med 2019;17(1):56.
link1
[332]
Alves VM, Golbraikh A, Capuzzi SJ, Liu K, Lam WI, Korn DR, et al. Multidescriptor read across (MuDRA): a simple and transparent approach for developing accurate quantitative structure‒activity relationship models. J Chem Inf Model 2018;58(6):1214‒23.
link1
[333]
Lei T, Chen F, Liu H, Sun H, Kang Y, Li D, et al. ADMET evaluation in drug discovery. Part 17: development of quantitative and qualitative prediction models for chemical-induced respiratory toxicity. Mol Pharm 2017;14(7):2407‒21.
link1
[334]
Zhu L, Zhao J, Zhang Y, Zhou W, Yin L, Wang Y, et al. ADME properties evaluation in drug discovery: in silico prediction of blood‒brain partitioning. Mol Divers 2018;22(4):979‒90.
link1
[335]
Su BH, Tu YS, Lin C, Shao CY, Lin OA, Tseng YJ. Rule-based prediction models of cytochrome P450 inhibition. J Chem Inf Model 2015;55(7):1426‒34.
link1
[336]
Yang M, Chen J, Xu L, Shi X, Zhou X, Xi Z, et al. A novel adaptive ensemble classification framework for ADME prediction. RSC Adv 2018;8(21):11661‒83.
link1
[337]
Radchenko EV, Dyabina AS, Palyulin VA. Towards deep neural network models for the prediction of the blood‒brain barrier permeability for diverse organic compounds. Molecules 2020;25(24):5901.
link1
[338]
Wang D, Liu W, Shen Z, Jiang L, Wang J, Li S, et al. Deep learning based drug metabolites prediction. Front Pharmacol 2020;10(1):1586.
link1
[339]
Yang H, Lou C, Sun L, Li J, Cai Y, Wang Z, et al. admetSAR 2.0: web-service for prediction and optimization of chemical ADMET properties. Bioinformatics 2019;35(6):1067‒9.
link1
[340]
Daina A, Michielin O, Zoete V. SwissADME: a free web tool to evaluate pharmacokinetics, drug-likeness and medicinal chemistry friendliness of small molecules. Sci Rep 2017;7(1):42717.
link1
[341]
Banerjee P, Eckert AO, Schrey AK, Preissner R. ProTox-II: a webserver for the prediction of toxicity of chemicals. Nucleic Acids Res 2018;46(W1): W257‒63.
link1
[342]
Pires DE, Blundell TL, Ascher DB. pkCSM: predicting small-molecule pharmacokinetic and toxicity properties using graph-based signatures. J Med Chem 2015;58(9):4066‒72.
link1
[343]
Hay M, Thomas DW, Craighead JL, Economides C, Rosenthal J. Clinical development success rates for investigational drugs. Nat Biotechnol 2014;32(1):40‒51.
link1
[344]
Harrer S, Shah P, Antony B, Hu J. Artificial intelligence for clinical trial design. Trends Pharmacol Sci 2019;40(8):577‒91.
link1
[345]
Perez-Gracia JL, Sanmamed MF, Bosch A, Patiño-Garcia A, Schalper KA, Segura V, et al. Strategies to design clinical studies to identify predictive biomarkers in cancer research. Cancer Treat Rev 2017;53(1):79‒97.
link1
[346]
Banda JM, Seneviratne M, Hernandez-Boussard T, Shah NH. Advances in electronic phenotyping: from rule-based definitions to machine learning models. Annu Rev Biomed Data Sci 2018;1(1):53‒68.
link1
[347]
Palmqvist S, Insel PS, Zetterberg H, Blennow K, Brix B, Stomrud E, et al.; Alzheimer’s Disease Neuroimaging Initiative, Swedish BioFINDER Study. Accurate risk estimation of β-amyloid positivity to identify prodromal Alzheimer’s disease: cross-validation study of practical algorithms. Alzheimers Dement 2019;15(2):194‒204.
link1
[348]
Romero K, Ito K, Rogers JA, Polhamus D, Qiu R, Stephenson D, et al. The future is now: model-based clinical trial design for Alzheimer’s disease. Clin Pharmacol Ther 2015;97(3):210‒4.
link1
[349]
Bain EE, Shafner L, Walling DP, Othman AA, Chuang-Stein C, Hinkle J, et al. Use of a novel artificial intelligence platform on mobile devices to assess dosing compliance in a phase 2 clinical trial in subjects with schizophrenia. JMIR Mhealth Uhealth 2017;5(2):e18.
link1
[350]
Yauney G, Shah P. Reinforcement learning with action-derived rewards for chemotherapy and clinical trial dosing regimen selection. In: Proceedings of the 3rd Machine Learning for Healthcare Conference; 2018 Aug 17‒18; Stanford, CA, USA; 2018. p. 161‒226.
[351]
Farrington CP. Relative incidence estimation from case series for vaccine safety evaluation. Biometrics 1995;51(1):228‒35.
link1
[352]
Ryan PB, Stang PE, Overhage JM, Suchard MA, Hartzema AG, DuMouchel W, et al. A comparison of the empirical performance of methods for a risk identification system. Drug Saf 2013;36(Suppl 1):143‒58.
link1
[353]
Norén GN, Hopstadius J, Bate A, Star K, Edwards IR. Temporal pattern discovery in longitudinal electronic patient records. Data Min Knowl Discov 2010;20(3):361‒87.
link1
[354]
Morel M, Bacry E, Gaïffas S, Guilloux A, Leroy F. ConvSCCS: convolutional selfcontrolled case series model for lagged adverse event detection. Biostatistics 2020;21(4):758‒74.
link1
[355]
Ben Abacha A, Chowdhury MFM, Karanasiou A, Mrabet Y, Lavelli A, Zweigenbaum P. Text mining for pharmacovigilance: using machine learning for drug name recognition and drug‒drug interaction extraction and classification. J Biomed Inform 2015;58(1):122‒32.
link1
[356]
Mower J, Subramanian D, Cohen T. Learning predictive models of drug sideeffect relationships from distributed representations of literature-derived semantic predications. J Am Med Inform Assoc 2018;25(10):1339‒50.
link1
[357]
Lorberbaum T, Nasir M, Keiser MJ, Vilar S, Hripcsak G, Tatonetti NP. Systems pharmacology augments drug safety surveillance. Clin Pharmacol Ther 2015;97(2):151‒8.
link1
[358]
Enshaei A, Robson CN, Edmondson RJ. Artificial intelligence systems as prognostic and predictive tools in ovarian cancer. Ann Surg Oncol 2015;22(12):3970‒5.
link1
[359]
Sun D, Wang M, Li A. A multimodal deep neural network for human breast cancer prognosis prediction by integrating multi-dimensional data. IEEE/ ACM Trans Comput Biol Bioinform 2018;16(3):841‒50.
link1
[360]
Chi CL, Street WN, Wolberg WH. Application of artificial neural networkbased survival analysis on two breast cancer datasets. AMIA Annu Symp Proc 2007;2007(1):130‒4.
[361]
Delen D, Walker G, Kadam A. Predicting breast cancer survivability: a comparison of three data mining methods. Artif Intell Med 2005;34(2):113‒27.
link1
[362]
Sun Y, Goodison S, Li J, Liu L, Farmerie W. Improved breast cancer prognosis through the combination of clinical and genetic markers. Bioinformatics 2007;23(1):30‒7.
link1
[363]
Gevaert O, De Smet F, Timmerman D, Moreau Y, De Moor B. Predicting the prognosis of breast cancer by integrating clinical and microarray data with Bayesian networks. Bioinformatics 2006;22(14):e184‒90.
link1
[364]
Lynch CM, Abdollahi B, Fuqua JD, de Carlo AR, Bartholomai JA, Balgemann RN, et al. Prediction of lung cancer patient survival via supervised machine learning classification techniques. Int J Med Inform 2017;108(1):1‒8.
link1
[365]
Yu KH, Zhang C, Berry GJ, Altman RB, Ré C, Rubin DL, et al. Predicting nonsmall cell lung cancer prognosis by fully automated microscopic pathology image features. Nat Commun 2016;7(1):12474.
link1
[366]
Biglarian A, Hajizadeh E, Kazemnejad A, Zali M. Application of artificial neural network in predicting the survival rate of gastric cancer patients. Iran J Public Health 2011;40(2):80‒6.
[367]
Zhu Y, Wang QC, Xu MD, Zhang Z, Cheng J, Zhong YS, et al. Application of convolutional neural network in the diagnosis of the invasion depth of gastric cancer based on conventional endoscopy. Gastrointest Endosc 2019;89(4):806‒15.
link1
[368]
Zhu L, Luo W, Su M, Wei H, Wei J, Zhang X, et al. Comparison between artificial neural network and Cox regression model in predicting the survival rate of gastric cancer patients. Biomed Rep 2013;1(5):757‒60.
link1
[369]
Tian DW, Wu ZL, Jiang LM, Gao J, Wu CL, Hu HL. Neural precursor cell expressed, developmentally downregulated 8 promotes tumor progression and predicts poor prognosis of patients with bladder cancer. Cancer Sci 2019;110(1):458‒67.
link1
[370]
Hasnain Z, Mason J, Gill K, Miranda G, Gill IS, Kuhn P, et al. Machine learning models for predicting post-cystectomy recurrence and survival in bladder cancer patients. PLoS One 2019;14(2):e0210976.
link1
[371]
Kuo RJ, Huang MH, Cheng WC, Lin CC, Wu YH. Application of a two-stage fuzzy neural network to a prostate cancer prognosis system. Artif Intell Med 2015;63(2):119‒33.
link1
[372]
Zhang S, Xu Y, Hui X, Yang F, Hu Y, Shao J, et al. Improvement in prediction of prostate cancer prognosis with somatic mutational signatures. J Cancer 2017;8(16):3261‒7.
link1
[373]
Corey EJ, Wipke WT. Computer-assisted design of complex organic syntheses. Science 1969;166(3902):178‒92.
link1
[374]
Struble TJ, Alvarez JC, Brown SP, Chytil M, Cisar J, DesJarlais RL, et al. Current and future roles of artificial intelligence in medicinal chemistry synthesis. J Med Chem 2020;63(16):8667‒82.
link1
[375]
Segler MHS, Preuss M, Waller MP. Planning chemical syntheses with deep neural networks and symbolic AI. Nature 2018;555(7698):604‒10.
link1
[376]
Gao H, Struble TJ, Coley CW, Wang Y, Green WH, Jensen KF. Using machine learning to predict suitable conditions for organic reactions. ACS Cent Sci 2018;4(11):1465‒76.
link1
[377]
Gong Y, Xue D, Chuai G, Yu J, Liu Q. DeepReac+: deep active learning for quantitative modeling of organic chemical reactions. Chem Sci 2021;12(43):14459‒72.
link1
[378]
Coley CW, Barzilay R, Jaakkola TS, Green WH, Jensen KF. Prediction of organic reaction outcomes using machine learning. ACS Cent Sci 2017;3(5):434‒43.
link1
[379]
Caramelli D, Salley D, Henson A, Camarasa GA, Sharabi S, Keenan G, et al. Networking chemical robots for reaction multitasking. Nat Commun 2018;9(1):3406.
link1
[380]
Merrifield RB. Automated synthesis of peptides. Science 1965;150(3693):178‒85.
link1
[381]
Alvarado-Urbina G, Sathe GM, Liu WC, Gillen MF, Duck PD, Bender R, et al. Automated synthesis of gene fragments. Science 1981;214(4518):270‒4.
link1
[382]
Doi T, Fuse S, Miyamoto S, Nakai K, Sasuga D, Takahashi T. A formal total synthesis of taxol aided by an automated synthesizer. Chem Asian J 2006;1(3):370‒83.
link1
[383]
Boström J, Brown DG, Young RJ, Keserü GM. Expanding the medicinal chemistry synthetic toolbox. Nat Rev Drug Discov 2018;17(10):709‒27. Erratum in: Nat Rev Drug Discov 2018;17(12):922.
link1
[384]
Bellomo A, Celebi-Olcum N, Bu X, Rivera N, Ruck RT, Welch CJ, et al. Rapid catalyst identification for the synthesis of the pyrimidinone core of HIV integrase inhibitors. Angew Chem Int Ed Engl 2012;51(28):6912‒5.
link1
[385]
Dreher SD, Dormer PG, Sandrock DL, Molander GA. Efficient cross-coupling of secondary alkyltrifluoroborates with aryl chlorides—reaction discovery using parallel microscale experimentation. J Am Chem Soc 2008;130(29):9257‒9.
link1
[386]
Buitrago Santanilla A, Regalado EL, Pereira T, Shevlin M, Bateman K, Campeau LC, et al. Nanomole-scale high-throughput chemistry for the synthesis of complex molecules. Science 2015;347(6217):49‒53.
link1
[387]
Perera D, Tucker JW, Brahmbhatt S, Helal CJ, Chong A, Farrell W, et al. A platform for automated nanomole-scale reaction screening and micromolescale synthesis in flow. Science 2018;359(6374):429‒34.
link1
[388]
Shevlin M. Practical high-throughput experimentation for chemists. ACS Med Chem Lett 2017;8(6):601‒7.
link1
[389]
Ahneman DT, Estrada JG, Lin S, Dreher SD, Doyle AG. Predicting reaction performance in C‒N cross-coupling using machine learning. Science 2018;360(6385):186‒90.
link1
[390]
Isayev O. Text mining facilitates materials discovery. Nature 2019;571(7763):42‒3.
link1
[391]
Lee J, Yoon W, Kim S, Kim D, Kim S, So CH, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text mining. Bioinformatics 2020;36(4):1234‒40.
link1
[392]
Sun C, Yang Z, Luo L, Wang L, Wang J. A deep learning approach with deep contextualized word representations for chemical‒protein interaction extraction from biomedical literature. IEEE Access 2019;7(1):151034‒46.
link1
[393]
Zhao S, Su C, Lu Z, Wang F. Recent advances in biomedical literature mining. Brief Bioinform 2021;22(3):bbaa057.
link1
[394]
Deftereos SN, Andronis C, Friedla EJ, Persidis A, Persidis A. Drug repurposing and adverse event prediction using high-throughput literature analysis. Wiley Interdiscip Rev Syst Biol Med 2011;3(3):323‒34.
link1
[395]
Yang HT, Ju JH, Wong YT, Shmulevich I, Chiang JH. Literature-based discovery of new candidates for drug repurposing. Brief Bioinform 2017;18(3):488‒97.
[396]
Zhang R, Cairelli MJ, Fiszman M, Kilicoglu H, Rindflesch TC, Pakhomov SV, et al. Exploiting literature-derived knowledge and semantics to identify potential prostate cancer drugs. Cancer Inform 2014;13(Suppl 1):103‒11.
link1
[397]
Hu Y, Hines LM, Weng H, Zuo D, Rivera M, Richardson A, et al. Analysis of genomic and proteomic data using advanced literature mining. J Proteome Res 2003;2(4):405‒12.
link1
[398]
Shang N, Xu H, Rindflesch TC, Cohen T. Identifying plausible adverse drug reactions using knowledge extracted from the literature. J Biomed Inform 2014;52(1):293‒310.
link1
[399]
Malec SA, Wei P, Bernstam EV, Boyce RD, Cohen T. Using computable knowledge mined from the literature to elucidate confounders for EHR-based pharmacovigilance. J Biomed Inform 2021;117(1):103719.
link1
[400]
Wang LL, Lo K. Text mining approaches for dealing with the rapidly expanding literature on COVID-19. Brief Bioinform 2021;22(2):781‒99.
link1
[401]
Feng Z, Shen Z, Li H, Li S. e-TSN: an interactive visual exploration platform for target‒disease knowledge mapping from literature. Brief Bioinform 2022;23(6):bbac465.
link1
[402]
Wang J, Shen Z, Liao Y, Yuan Z, Li S, He G, et al. Multi-modal chemical information reconstruction from images and texts for exploring the neardrug space. Brief Bioinform 2022;23(6):bbac461.
link1
[403]
Ahn AC, Tewari M, Poon CS, Phillips RS. The limits of reductionism in medicine: could systems biology offer an alternative? PLoS Med 2006;3(6): e208.
link1
[404]
König IR, Fuchs O, Hansen G, von Mutius E, Kopp MV. What is precision medicine? Eur Respir J 2017;50(4):1700391.
link1
[405]
Antman EM, Loscalzo J. Precision medicine in cardiology. Nat Rev Cardiol 2016;13(10):591‒602.
link1
[406]
Hingorani AD, van der Windt DA, Riley RD, Abrams K, Moons KGM, Steyerberg EW, et al. Prognosis research strategy (PROGRESS) 4: stratified medicine research. BMJ 2013;346:e5793.
link1
[407]
Tang J, Mou M, Wang Y, Luo Y, Zhu F. MetaFS: performance assessment of biomarker discovery in metaproteomics. Brief Bioinform 2021;22(3): bbaa105.
link1
[408]
Yang Q, Li B, Chen S, Tang J, Li Y, Li Y, et al. MMEASE: online meta-analysis of metabolomic data by enhanced metabolite annotation, marker selection and enrichment analysis. J Proteomics 2021;232(1):104023.
link1
[409]
Zhao Y, Pan Z, Namburi S, Pattison A, Posner A, Balachander S, et al. CUP-AIDx: a tool for inferring cancer tissue of origin and molecular subtype using RNA gene-expression data and artificial intelligence. EBioMedicine 2020;61(1):103030.
link1
[410]
Yeh YL, Su MW, Chiang BL, Yang YH, Tsai CH, Lee YL. Genetic profiles of transcriptomic clusters of childhood asthma determine specific severe subtype. Clin Exp Allergy 2018;48(9):1164‒72.
link1
[411]
Rolland DCM, Basrur V, Jeon YK, McNeil-Schwalm C, Fermin D, Conlon KP, et al. Functional proteogenomics reveals biomarkers and therapeutic targets in lymphomas. Proc Natl Acad Sci USA 2017;114(25):6581‒6.
link1
[412]
Niu L, Thiele M, Geyer PE, Rasmussen DN, Webel HE, Santos A, et al. Noninvasive proteomic biomarkers for alcohol-related liver disease. Nat Med 2022;28(6):1277‒87.
link1
[413]
Poon W, Kingston BR, Ouyang B, Ngo W, Chan WCW. A framework for designing delivery systems. Nat Nanotechnol 2020;15(10):819‒29.
link1
[414]
Mitchell MJ, Billingsley MM, Haley RM, Wechsler ME, Peppas NA, Langer R. Engineering precision nanoparticles for drug delivery. Nat Rev Drug Discov 2021;20(2):101‒24.
link1
[415]
Li J, Esteban-Fernández de Ávila B, Gao W, Zhang L, Wang J. Micro/nanorobots for biomedicine: delivery, surgery, sensing, and detoxification. Sci Robot 2017;2(4):eaam6431.
link1
[416]
Ong AT, Serruys PW. Technology insight: an overview of research in drugeluting stents. Nat Clin Pract Cardiovasc Med 2005;2(12):647‒58.
link1
[417]
Bhatia SN, Chen X, Dobrovolskaia MA, Lammers T. Cancer nanomedicine. Nat Rev Cancer 2022;22(10):550‒6.
link1
[418]
Vamathevan J, Clark D, Czodrowski P, Dunham I, Ferran E, Lee G, et al. Applications of machine learning in drug discovery and development. Nat Rev Drug Discov 2019;18(6):463‒77.
link1
[419]
Ekins S, Puhl AC, Zorn KM, Lane TR, Russo DP, Klein JJ, et al. Exploiting machine learning for end-to-end drug discovery and development. Nat Mater 2019;18(5):435‒41.
link1
[420]
Chen C, Yaari Z, Apfelbaum E, Grodzinski P, Shamay Y, Heller DA. Merging data curation and machine learning to improve nanomedicines. Adv Drug Deliv Rev 2022;183(1):114172.
link1
[421]
Reker D, Rybakova Y, Kirtane AR, Cao R, Yang JW, Navamajiti N, et al. Computationally guided high-throughput design of self-assembling drug nanoparticles. Nat Nanotechnol 2021;16(6):725‒33.
link1
[422]
Shamay Y, Shah J, Is_ık M, Mizrachi A, Leibold J, Tschaharganeh DF, et al. Quantitative self-assembly prediction yields targeted nanomedicines. Nat Mater 2018;17(4):361‒8.
link1
[423]
Lu Y, Aimetti AA, Langer R, Gu Z. Bioresponsive materials. Nat Rev Mater 2016;2(1):16075.
link1
[424]
Santana R, Zuluaga R, Gañán P, Arrasate S, Onieva E, González-Díaz H. Predicting coated-nanoparticle drug release systems with perturbationtheory machine learning (PTML) models. Nanoscale 2020;12(25):13471‒83.
link1
[425]
Owh C, Ow V, Lin Q, Wong JHM, Ho D, Loh XJ, et al. Bottom-up design of hydrogels for programmable drug release. Biomater Adv 2022;141 (1):213100.
link1
[426]
Boztepe C, Künkül A, Yüceer M. Application of artificial intelligence in modeling of the doxorubicin release behavior of pH and temperature responsive poly (NIPAAm-co-AAc)-PEG IPN hydrogel. J Drug Deliv Sci Technol 2020;57(1):101603.
link1
[427]
Stiepel RT, Pena ES, Ehrenzeller SA, Gallovic MD, Lifshits LM, Genito CJ, et al. A predictive mechanistic model of drug release from surface eroding polymeric nanoparticles. J Control Release 2022;351(1):883‒95.
link1
[428]
Jayatunga MKP, Xie W, Ruder L, Schulze U, Meier C. AI in small-molecule drug discovery: a coming wave? Nat Rev Drug Discov 2022;21(3):175‒6.
link1
[429]
Richardson P, Griffin I, Tucker C, Smith D, Oechsle O, Phelan A, et al. Baricitinib as potential treatment for 2019-nCoV acute respiratory disease. Lancet 2020;395(10223):e30‒1.
link1
[430]
Kirkpatrick P. Artificial intelligence makes a splash in small-molecule drug discovery. Biopharm Deal 2022;16(2):84‒6.
[431]
Zhang C, Mou M, Zhou Y, Zhang W, Lian X, Shi S, et al. Biological activities of drug inactive ingredients. Brief Bioinform 2022;23(5):bbac160.
link1
[432]
Haghverdi L, Lun ATL, Morgan MD, Marioni JC. Batch effects in single-cell RNA-sequencing data are corrected by matching mutual nearest neighbors. Nat Biotechnol 2018;36(5):421‒7.
link1













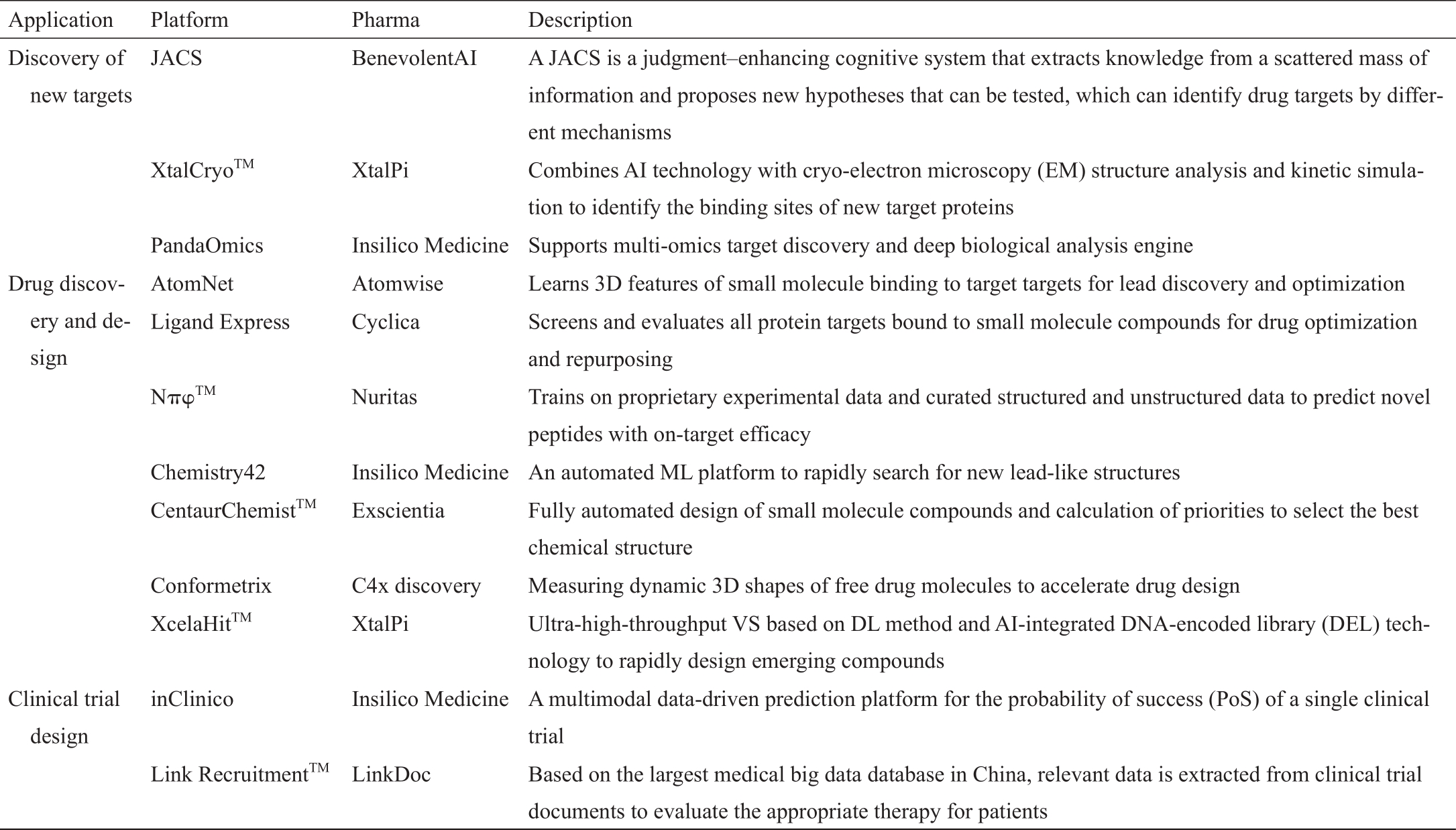











 京公网安备 11010502051620号
京公网安备 11010502051620号




